Kit Perpustakaan TIANSeq DirectFast (illumina)
Fitur
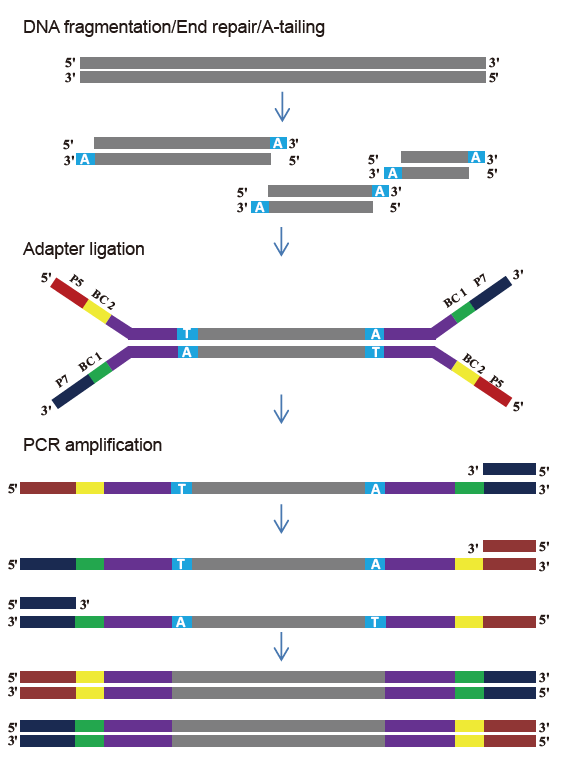
■ Seragam urutan sing apik: Ora ana bias dhasar proses fragmentasi DNA lan proses amplifikasi PCR.
■ Efisiensi konversi perpustakaan sing dhuwur: konstruksi perpustakaan kanthi efisiensi dhuwur bisa dipesthekake kanggo sampel DNA 1 ng.
■ Operasi cepet: Kabeh proses pambangunan perpustakaan mung butuh 2,5 jam.
■ Hemat biaya: Ora perlu instrumen lan peralatan khusus。
Spesifikasi
Jinis: Preparasi perpustakaan DNA kanggo platform urutan urutan dhuwur saka illumina
Conto: DNA genomik utawa DNA fragmen gedhe
Target: DNA kaping pindho
Miwiti conto input: 1 ng- 1 μg
Wektu operasi: 2,5 jam
Aplikasi hilir: Urutan ing platform illumina
Kabeh produk bisa disesuaikan kanggo ODM / OEM. Kanggo rincian,monggo klik Layanan Khusus (ODM / OEM)
Input sampel sing fleksibel lan ukuran fragmen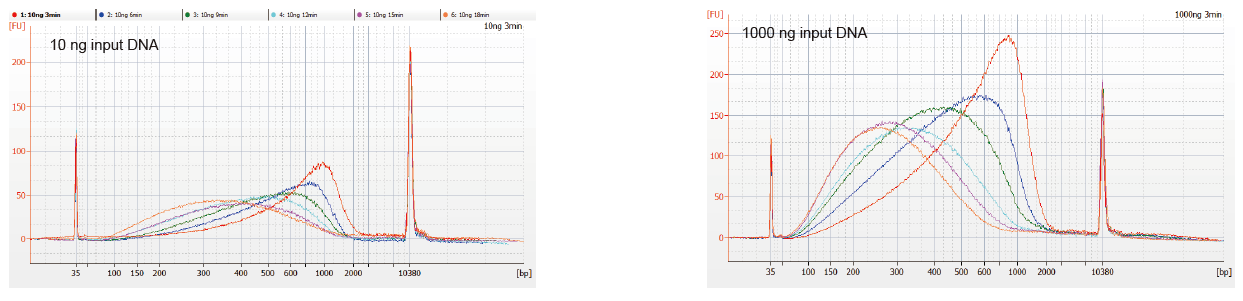 |
Gambar 1. Profil fragmen DNA kanthi reaksi reaksi sing beda. 10 ng lan 1000 ng DNA dipecah dadi nggunakake TIANSeq DirectFast DNA Library Kit. Produk reaksi sing diobati kanthi beda wektu reaksi diresiki manik-manik magnetik 1,8 × Ampure XP lan dianalisis Angilent 2100. |
Jangkoan Urutan Kaya Covaris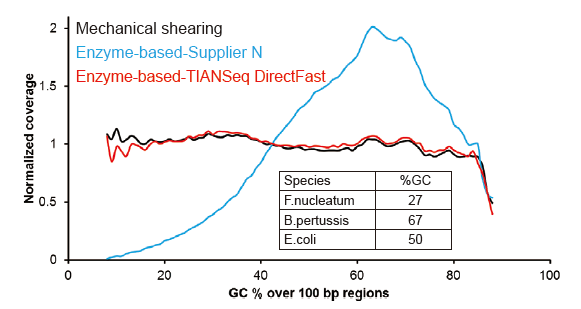 |
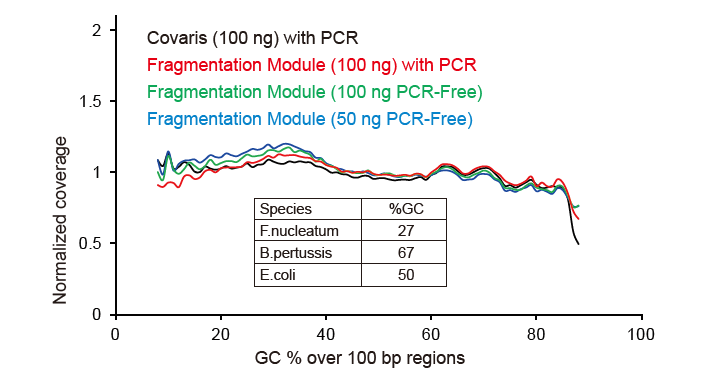
Gambar 2. Perbandingan cakupan genom kanthi cara nyiyapake perpustakaan sing beda. Telung DNA genomik bakteri kanthi isi GC sing beda-beda kalebu ekuimolar campuran, lan asil liputan genom saka 100 ng perpustakaan DNA campuran nggunakake metode kasebut dibandhingake. Asil nuduhake manawa Kit Perpustakaan TIANSeq DirectFast nduwe pengaruh sing padha marang fragmentasi DNA kaya gunting mekanik, lan ora ana bias dhasar fragmentasi. |
Ora Ana Bias Sistematik sing Paling Kurang 1 Ng input DNA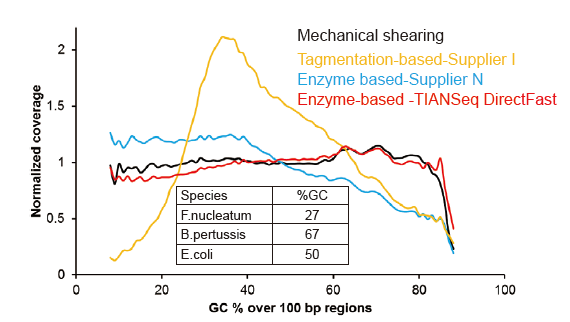 |
Gambar 3. Perbandingan cakupan genom kanthi cara nyiyapake perpustakaan sing beda. Telung DNA genomik bakteri kanthi isi GC sing beda-beda kalebu ekuimolar campuran, lan asil liputan genom saka 1 ng perpustakaan DNA campuran nggunakake metode kasebut dibandhingake. Asil nuduhake manawa TIANSeq DirectFast Library Kit duwe efek fragmentasi sing konsisten karo gunting mekanik sanajan input DNA kurang saka 1 ng, lan ora ana bias dhasar. |
| Bisa Aliran Kerja Bebas PCR
|
Gambar 4. Input DNA genom sing beda digunakake kanggo mbangun perpustakaan kanthi konstruksi PCR utawa PCR bebas, lan asil jangkoan genom dibandhingake. Asil nuduhake manawa kanthi operasi siji-tabung lan langkah-langkah pambangunan perpustakaan sing efisien, perpustakaan DNA sing dibangun karo TIANSeq DirectFast Library Kit njaga konsistensi sing dhuwur karo geser mekanik ing distribusi jangkoan urutan fragmen kanggo alur kerja PCR tanpa pengayaan PCR. |
Statistik Efisiensi Konstruksi Perpustakaan lan Hasil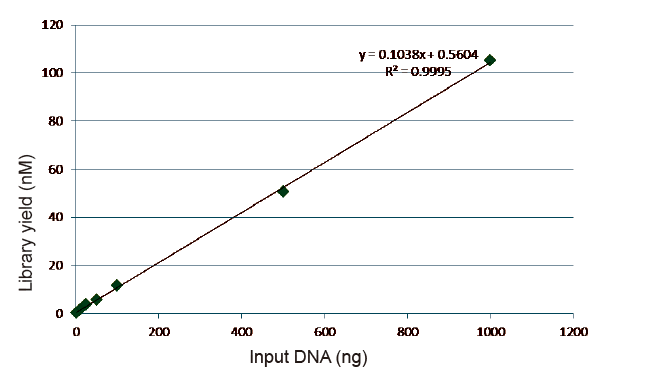 |
Gambar 5. Asil analisis kuantitatif DNA perpustakaan sing dipikolehi dening qPCR sawise konstruksi perpustakaan kanthi metode bebas PCR kanggo conto kanthi jumlah wiwitan sing beda (1, 10, 25, 50, 100, 500,1000 ng). Analisis regresi linier nunjukake manawa pangasilan perpustakaan duwe hubungan linier sing apik ing kisaran input sampel sing jembar. Kanggo input DNA sing kurang saka 1 ng, efisiensi konstruksi perpustakaan ora mudhun. |
Perbandingan Data Urutan Produk sing Beda
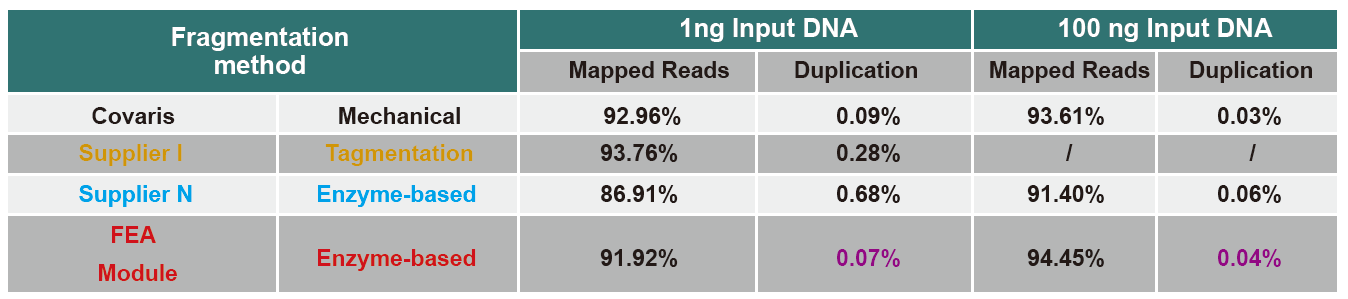
Saiki, teknologi urutan urutan-output dhuwur utamane adhedhasar teknologi sekuensing generasi sabanjure. Amarga dawa maca teknologi urutan sabanjure bisa diwatesi, kita kudu ngrampungake urutan dawa dadi perpustakaan fragmen cilik dadi urutan. Miturut kabutuhan eksperimen urutan sing beda-beda, biasane milih urutan urutan siji utawa urutan kaping pindho. Saiki fragmen DNA perpustakaan urutan sabanjure bakal disebar ing kisaran 200-800 bp.

a) DNA ora duwe kualitas lan ngemot penghambat. Gunakake conto DNA bermutu supaya ora nyegah aktivitas enzim.
b) Jumlah sampel DNA ora cukup nalika nggunakake metode bebas PCR kanggo mbangun perpustakaan DNA. Nalika input DNA fragmentine ngluwihi 50 ng, alur kerja bebas PCR bisa dipilih kanthi selektif sajrone proses konstruksi perpustakaan. Yen nomer salinan perpustakaan banget sithik supaya ora langsung diurutake, perpustakaan DNA bisa ditambah karo PCR sawise ligasi adaptor.
c) Kontaminasi RNA nyebabake kontaminasi DNA awal sing ora akurat Kontaminasi RNA bisa uga ana ing proses pemurnian DNA genom, sing bisa nyebabake kuantifikasi DNA sing akurat lan ngemot DNA sing kurang sajrone konstruksi perpustakaan. RNA bisa dicopot kanthi ngobati RNase.
A-1
a) Perangan cilik (60 bp-120 bp) katon Perangan cilik biasane fragmen adaptor utawa dimensi sing digawe adaptor. Pemurnian nganggo manik magnetik Agencourt AMPure XP kanthi efektif bisa ngilangi fragmen adaptor kasebut lan njamin kualitas urutan.
b) Fragmen gedhe katon ing perpustakaan sawise amplifikasi PCR Ukuran fragmen DNA perpustakaan bakal nambah 120 bp sawise adaptor ligasi. Yen fragmen DNA mundhak luwih saka 120 bp sawise ligasi adaptor, bisa uga disebabake amplifikasi fragmen abnormal saka amplifikasi PCR sing gedhe banget. Nyuda jumlah siklus PCR bisa nyegah kahanan kasebut.
c) Ukuran fragmen DNA perpustakaan sing ora normal sawise ligasi adaptor Dawane adaptor ing kit iki yaiku 60 bp. Nalika rong ujung fragmen digandhengake karo adaptor, dawane mung bakal nambah 120 bp. Nalika nggunakake adaptor liyane saka sing diwenehake karo kit iki, hubungi supplier kanggo menehi informasi sing relevan kayata dawa adaptor. Priksa manawa alur kerja lan operasi eksperimen tindakake langkah-langkah sing diterangake ing manual.
d) Ukuran fragmen DNA sing ora normal sadurunge ligation adaptor Alesan kanggo masalah iki bisa uga disebabake dening kahanan reaksi sing salah sajrone fragmen DNA. Wektu reaksi sing beda kudu digunakake kanggo input DNA sing beda. Yen input DNA luwih saka 10 ng, disaranake milih wektu reaksi 12 menit minangka wektu wiwitan kanggo ngoptimalake, lan ukuran fragmen sing diprodhuksi ing wektu iki utamane ing kisaran 300-500 bp. Pangguna bisa nambah utawa nyuda dawa fragmen DNA nganti 2-4 menit miturut sarat dhewe kanggo ngoptimalake fragmen DNA kanthi ukuran sing dibutuhake.
A-2
a) Wektu fragmen ora dioptimalake Yen DNA fragmen cilik banget utawa gedhe banget, waca Pedoman Pilihan Wektu Fragmentasi sing diwenehake ing pandhuan kanggo nemtokake wektu reaksi, lan gunakake titik wektu iki minangka kontrol, Kajaba iku nyiyapake sistem reaksi kanggo ndawakake utawa nyepetake 3 menit supaya bisa nyetel wektu fragmentasi sing luwih akurat.
A-3
Distribusi ukuran DNA sing normal sawise perawatan fragmentasi
a) Cara pambocoran reagen fragmentasi sing salah, utawa reagen ora dicampur sawise dicair. Nglereni reagen campuran 5 × Fragmentasi ing es. Sawise dicair, dicampur reagen kanthi merata kanthi alon-alon gulung ing ngisor tabung. Aja vorteks reagen!
b) Sampel input DNA ngemot EDTA utawa polutan liyane Panipisan ion uyah lan agen chelating ing langkah pemurnian DNA penting banget kanggo sukses eksperimen kasebut. Yen DNA dibubarake ing 1 × TE, gunakake cara sing diwenehake kanggo instruksi nindakake fragmen. Yen konsentrasi EDTA ing larutan kasebut durung mesthi, disaranake ngresiki DNA lan larut ing banyu sing deionisasi kanggo reaksi sabanjure.
c) Kualifikasi DNA awal sing ora akurat Ukuran DNA fragmen gegandhengan banget karo jumlah input DNA. Sadurunge perawatan fragmentasi, kuantifikasi DNA sing akurat nggunakake Qubit, Picogreen lan metode liyane penting kanggo nemtokake jumlah DNA sing pas ing sistem reaksi.
d) Preparasi sistem reaksi ora nuruti prentah Persiyapan sistem reaksi fragmen kudu ditindakake ing es kanthi manut paugeran. Kanggo mesthekake efek sing paling apik, kabeh komponen reaksi kudu dilebokake ing es lan persiyapan sistem reaksi kudu ditindakake sawise adhem lengkap. Sawise persiyapan rampung, coba gulung cepet utawa pipet supaya dicampur kanthi rapi. Aja pusaran!
1. Metode campuran sing ora cocog (vorteks, osilasi kasar, lsp) bakal nyebabake distribusi fragmen perpustakaan sing ora normal (kaya sing dituduhake ing gambar ing ngisor iki), saengga bisa nyebabake kualitas perpustakaan. Mula, nalika nyiyapake solusi reaksi Campuran Fragmentasi, mangga pipet munggah lan mudhun kanthi alon-alon kanggo dicampur, utawa gunakake pucuk driji kanggo gulung cepet lan campuran kanthi rata. Ati-ati aja dicampur karo pusaran.

2. DNA kemurnian dhuwur kudu digunakake kanggo pambangunan perpustakaan
■ Integritas DNA sing apik: Pita elektroforesis luwih saka 30 kb, tanpa buntut
■ OD260 / 230:> 1.5
■ OD260 / 280: 1.7-1.9
3. Jumlah input DNA kudu akurat Disaranake nggunakake metode Qubit lan PicoGreen kanggo ngetung DNA, tinimbang Nanodrop.
4. Isi EDTA ing larutan DNA kudu ditemtokake EDTA duwe pengaruhe gedhe tumrap reaksi fragmentasi. Yen isi EDTA dhuwur, pemurnian DNA kudu ditindakake sadurunge tes sabanjure.
5. Solusi reaksi fragmentasi kudu disiapake ing es. Proses fragmentasi sensitif karo suhu lan wektu reaksi (utamane sawise nambah penambah). Kanggo mesthekake akurasi wektu reaksi, mangga nyiyapake sistem reaksi ing es.
6. Wektu reaksi fragmentasi kudu akurat Wektu reaksi langkah fragmentasi bakal langsung mengaruhi ukuran produk fragmen, mula mengaruhi distribusi ukuran fragmen DNA ing perpustakaan.
1. Apa jinis sampel sing ditrapake kanggo kit iki?
Jinis conto sing cocog kanggo kit iki bisa kalebu RNA total utawa mRNA murni kanthi integritas RNA sing apik. Yen RNA total digunakake kanggo mbangun perpustakaan, disaranake nggunakake kit depresion rRNA (Cat # 4992363/4992364/4992391) kanggo mbusak rRNA luwih dhisik.
2. Apa conto FFPE bisa digunakake kanggo mbangun perpustakaan nganggo kit iki?
MRNA ing conto FFPE bakal mudhun nganti sawetara level, kanthi integritas relatif kurang. Nalika nggunakake kit iki kanggo pambangunan perpustakaan, disaranake ngoptimalake wektu fragmentasi (nyepetake wektu fragmentasi utawa ora nindakake fragmen).
3. Nggunakake langkah pilihan ukuran sing diwenehake ing manual produk, apa sing nyebabake segmen sing dipasang katon rada nyimpang?
Pilihan ukuran kudu ditindakake kanthi ketat miturut langkah pilihan ukuran ing manual produk iki. Yen ana penyimpangan, alasane manik-manik magnetik ora seimbang karo suhu ruangan utawa ora dicampur kabeh, pipet ora akurat utawa cairan tetep ana ing pucuk. Disaranake nggunakake tips kanthi adsorpsi sing sithik kanggo eksperimen kasebut.
4. Pilihan adaptor ing konstruksi perpustakaan
Kit konstruksi perpustakaan ora ngemot reagen adaptor, lan disaranake nggunakake kit iki bebarengan karo TIANSeq Single-Index Adapter (Illumina) (4992641/4992642/4992378).
5. QC perpustakaan
Deteksi kuantitatif perpustakaan: Qubit lan qPCR digunakake kanggo nemtokake konsentrasi massa lan konsentrasi molar perpustakaan. Operasi kasebut jumbuh banget karo manual produk. Konsentrasi perpustakaan umume memenuhi persyaratan urutan NGS. Deteksi kisaran distribusi perpustakaan: Nggunakake Agilent 2100 Bioanalyzer kanggo ndeteksi kisaran distribusi perpustakaan.
6. Pilihan nomer siklus amplifikasi
Miturut pandhuan, jumlah siklus PCR yaiku 6-12, lan jumlah siklus PCR sing dibutuhake kudu dipilih miturut input sampel. Ing perpustakaan asil dhuwur, over amplifikasi biasane ana ing macem-macem derajat, sing diwujudake kanthi pucuk sing rada gedhe sawise pucuk kisaran target ing deteksi Agilent 2100 Bioanalyzer, utawa konsentrasi Qubit sing dideteksi luwih murah tinimbang qPCR. Ringan saka amplifikasi minangka fenomena normal, sing ora mengaruhi urutan perpustakaan lan analisis data sabanjure.
7. Pucuk katon ing profil deteksi Agilent 2100 Bioanalyzer
Muncul paku ing deteksi Agilent 2100 Bioanalyzer amarga fragmen sampel sing ora rata, ing endi bakal ana fragmen liyane kanthi ukuran tartamtu, lan iki bakal dadi luwih jelas sawise pengayaan PCR. Ing kasus iki, disaranake ora nindakake pilihan ukuran, yaiku nyetel kondhisi fragmentasi dadi 94 ° C sajrone 15 menit inkubasi, ing endi distribusi fragmen cilik lan konsentrasi, lan homogenitas bisa ditambah.
Kategori produk
Napa PILIH KAMI
Wiwit didegake, pabrik kita wis ngembangake produk kelas kapisan kanthi ngetrapake prinsip
kualitas dhisik. Produk kita wis entuk reputasi apik ing industri lan valuabletrusty ing antarane para pelanggan anyar lan lawas ..